Uso de la herramienta Taxon Match del World Register of Marine Species - (WoRMS)
Published:
- En este post explicaré cómo usar la herramienta Taxon Match del World Register of Marine Species - WORMS, una herramienta automática para verificar la validez y la correcta escritura de los nombres de las especies, basada en el código APHIA.
¿Que es el World Register of Marine Species (WoRMS)?
El Registro Mundial de Especies Marinas (WoRMS por sus siglas en inglés) como su sitio web lo indica, “Es una lista autorizada y completa de nombres de organismos marinos que es mantenida por expertos taxónomos. WoRMS tiene un sistema de gestión editorial donde cada grupo taxonómico es representado por un experto que tiene la autoridad sobre el contenido y es responsable de controlar la calidad de la información. Cada uno de estos editores taxonómicos principales puede invitar a varios especialistas de grupos más pequeños dentro de su área de responsabilidad para que se unan a ellos”.
WoRMS es una base de datos taxonómica basada en una revisión por expertos. que tiene más de 200 editores, un equipo de gestión de datos y está basada en estándares internacionales.
La clasificación de los taxa superiores del WoRMS está basada en el artículo de Ruggiero et al. (2015), el cual los autores presentan “una clasificación de consenso de la vida para abarcar las más de 1,6 millones de especies ya proporcionada por más de 3.000 opiniones expertas de taxónomos en una forma unificada y coherente, sistema clasificado jerárquicamente conocido como Catalog of Life (CoL)”, esta clasificación va desde el Superreino hasta el nivel de Orden. Debajo de esta clasificación, los otros niveles taxonómicos (e.g. suboreden, familia, género, especie) son mantenidos por los editores de cada taxón.
¿Qué es el Taxon Match?
Dentro de este sitio existe una herramienta que sirve para revisar de forma automática la clasificación taxonómica de un listado propio de especies o taxa, desde un archivo de texto (.txt), un archivo separado por comas (.csv) o una tabla de Excel (.xls, .xlsx). Esta herramienta se conoce como el “Taxon Match”. El objetivo principal de la herramienta en hacer coincidir por medio de un algoritmo (TAXAMATCH fuzzy matching algorithm by Tony Rees) el listados de nombres que están en archivo, con los códigos Aphia, los nombres científicos válidos, las autoridades y otras salidas que son seleccionadas por el usuario. El límite máximo de entradas que se puede cargar por archivo es de 1500 filas.
¿Cómo usar el Taxon Match?
Cargar los datos y configurar la consulta.
La primera etapa es configurar todos los requerimientos para la consulta.
El primer paso que hay que hacer es preparar nuestro archivo con el listado de especies. En este tipo de archivo se debe tratar de incluir la mayor información posible con respecto a cada taxa que se trate de evaluar. Es recomendable que se incluyan columnas con datos como Familia, Género y Especie.
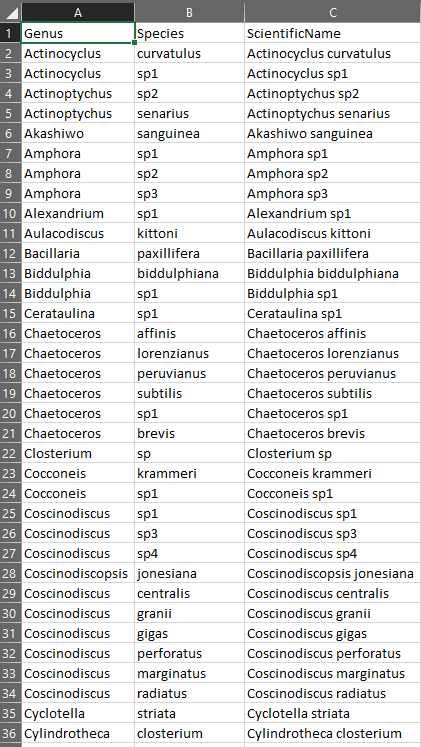
Es recomentable remover todas las notaciones que no hacen parte del nombre de la especie como pueden ser e.g. sp, cfr., larvae, indent, etc.
El segundo paso es seleccionar el archivo de la lista de especies y subirlo a través del botón “Examinar” ó “Choose File”. Esto abrirá un explorador de archivos que irá directamente a las carpetas del PC.
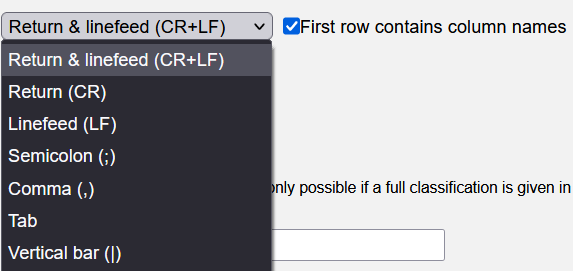
El tercer paso es seleccionar el tipo de delimitador de filas que se usó en el archivo del listado de especies e indicar si la primera fila contiene los nombres de cada una de las columnas o no.
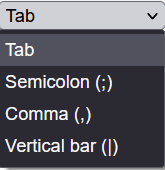
El cuarto paso es seleccionar el delimitador de tabulaciones que se ha usado para crear el archivo del listado de especie. Por ejemplo, si se usó una tabla de excel, por lo general se utiliza como delimitador de tabulaciones Tab, o si se ha creado un archivo .csv (Comma Separated Values) el separador de listas que se usa en este tipo de archivos pueden ser “;” o “,” dependiendo de cómo se tenga configurado el sistema operativo. Otra opción de delimitador de tabulaciones es la barra vertical que puede configurarse en algunos archivos de texto como un separador de tabulaciones.
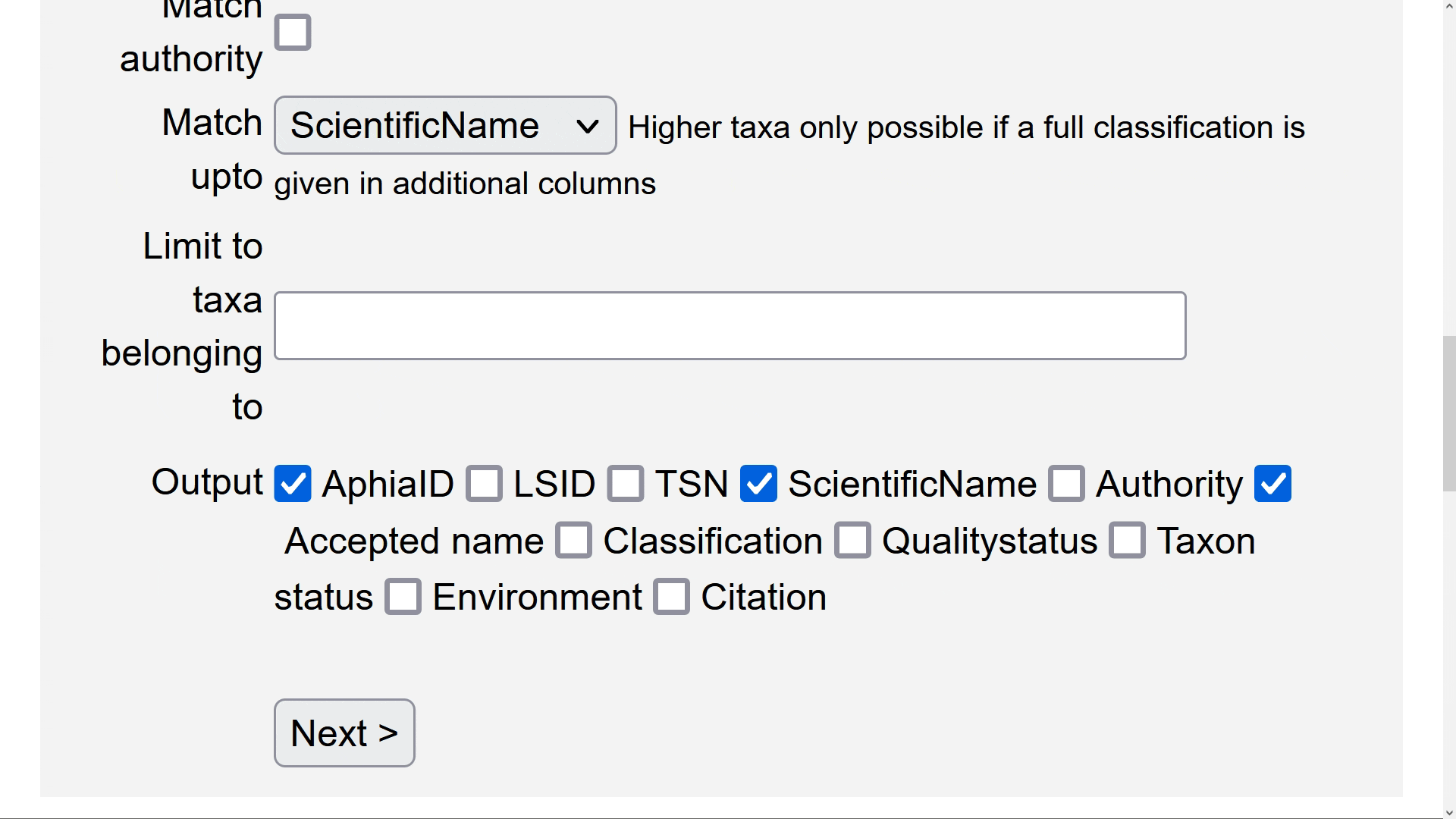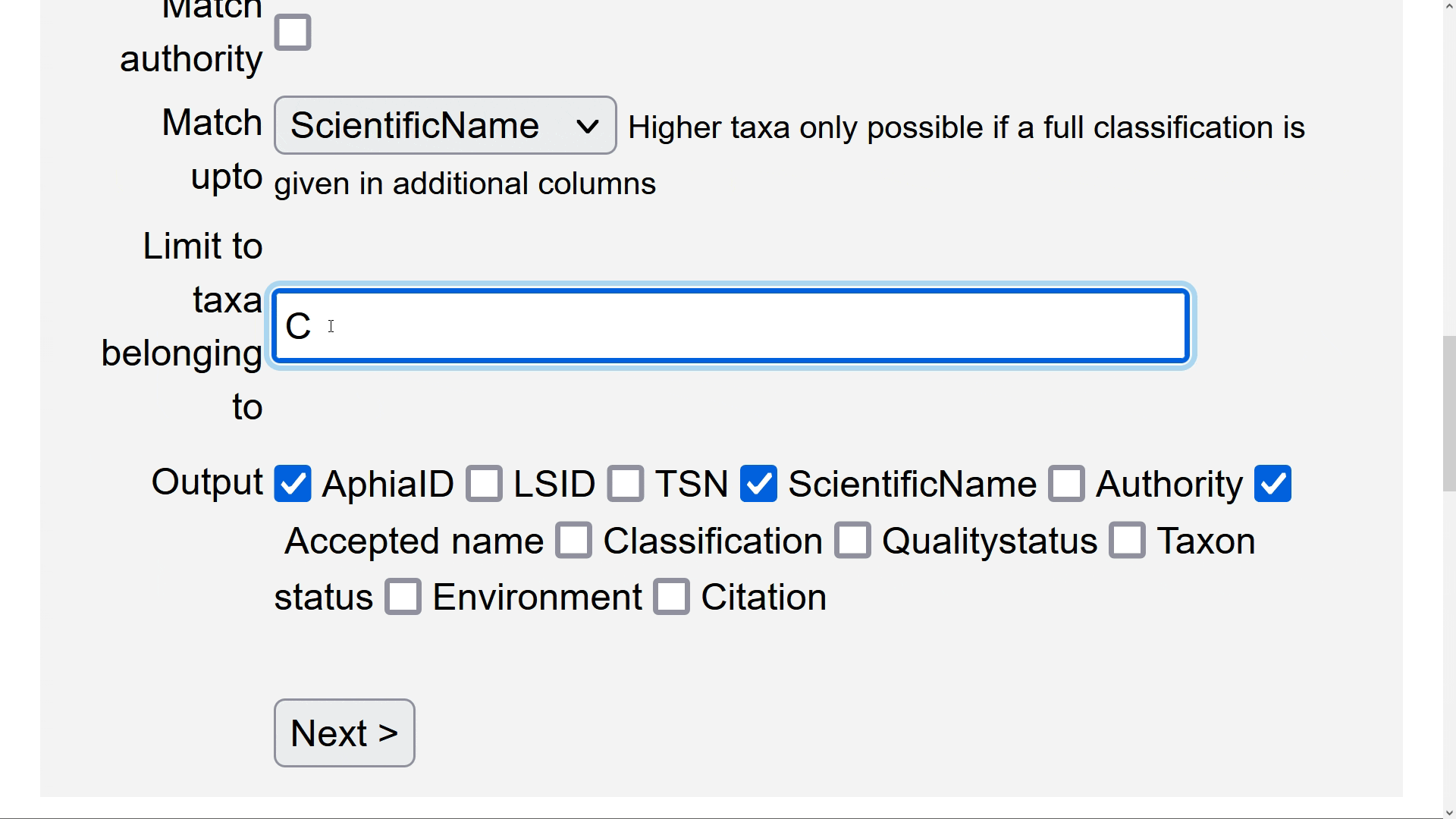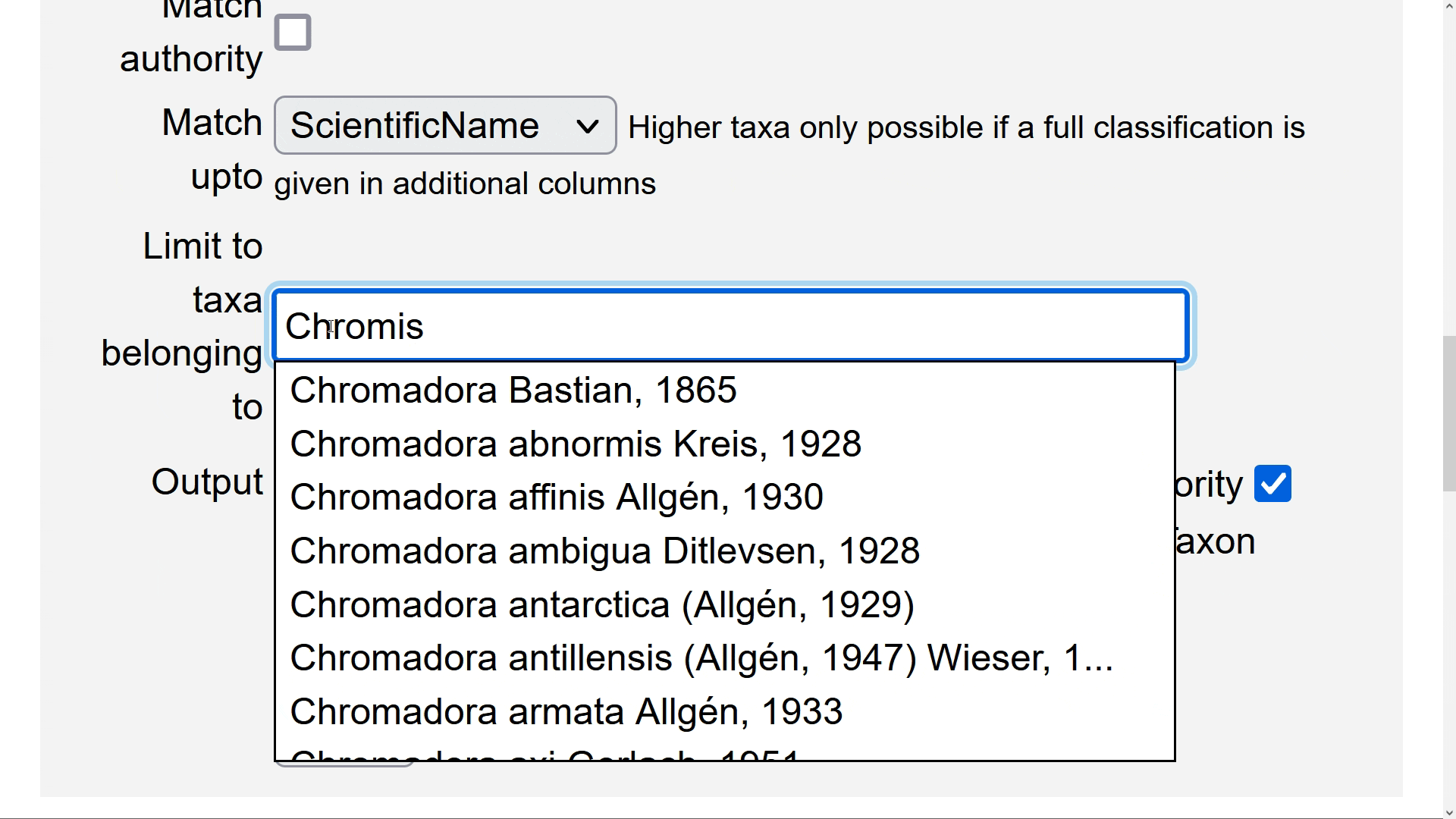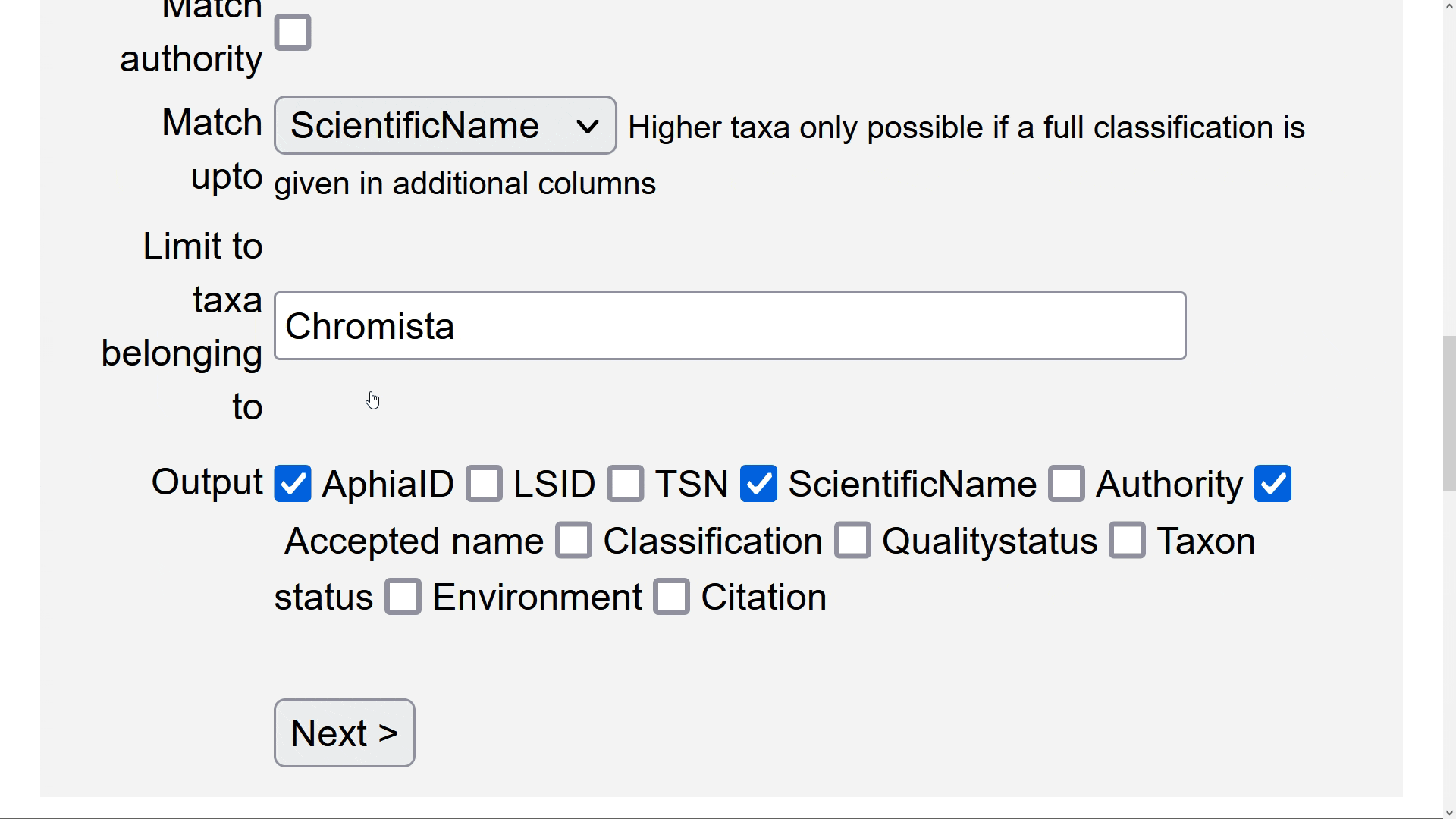
El quinto paso es la selección del botón de chequeo “Match authority” cuando se tiene una columna específica donde están los datos de autoridad del taxón y se desea evaluar una coincidencia para este dato.
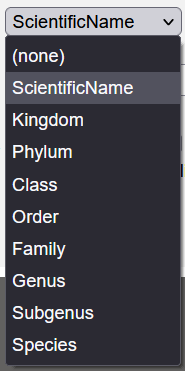
El sexto paso es decidir hasta qué nivel se desea coincidir el listado. Esto solo es posible si se han especificado columnas con una clasificación propia y esta se quiere evaluar a través de la herramienta. Si solo se tienen columnas con los datos de género especies y nombre científico, es preferible dejarlo por defecto con “ScientificName”.
El septimo paso es decidir si las coincidencias en la lista se limitan a un taxón superior al género exclusivamente (e.g. Porifera, Chromista), el cual se puede escribir y escoger de la posterior lista que se auto despliega en la entrada llamada Limit to taxa belonging to.
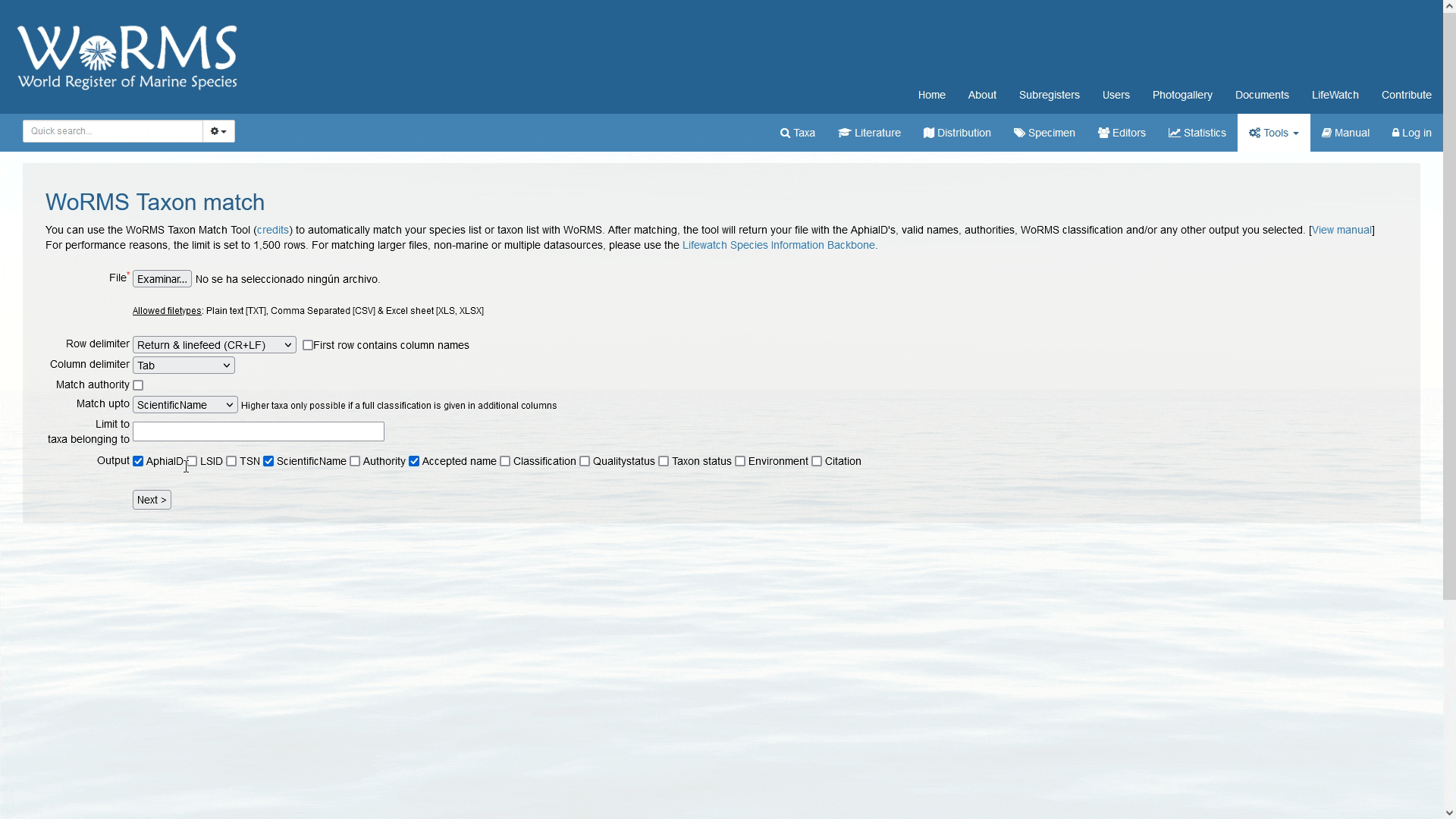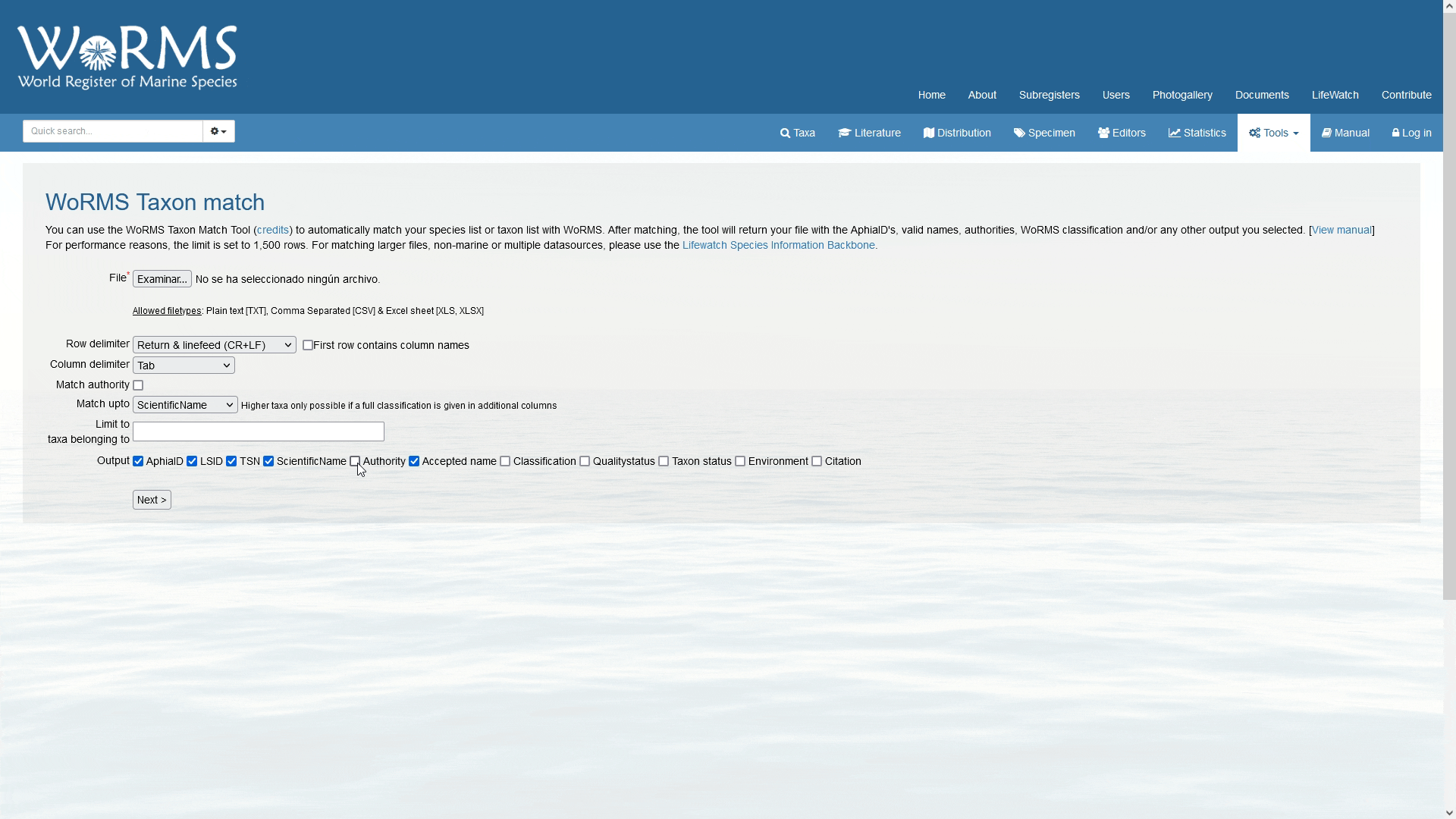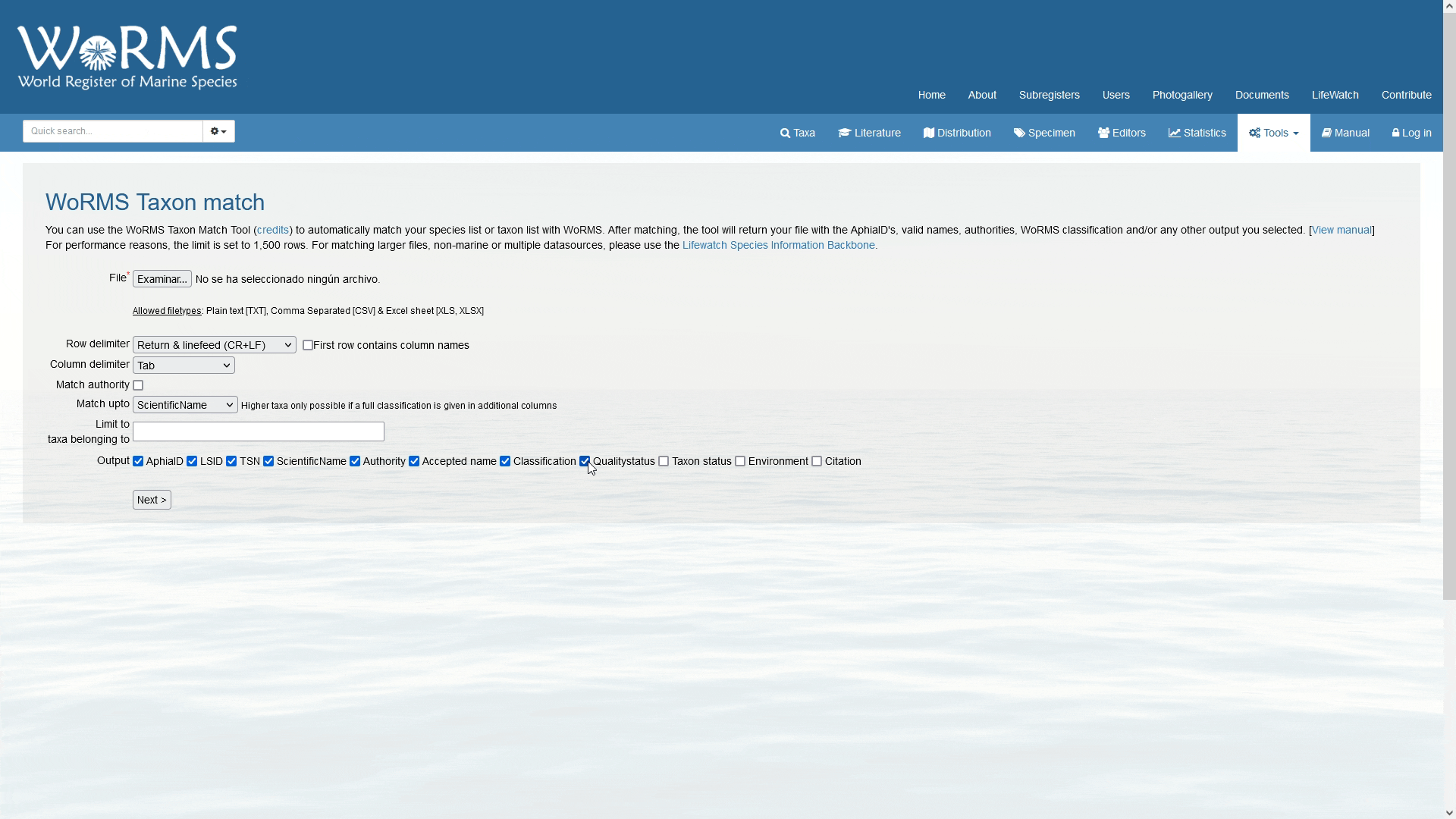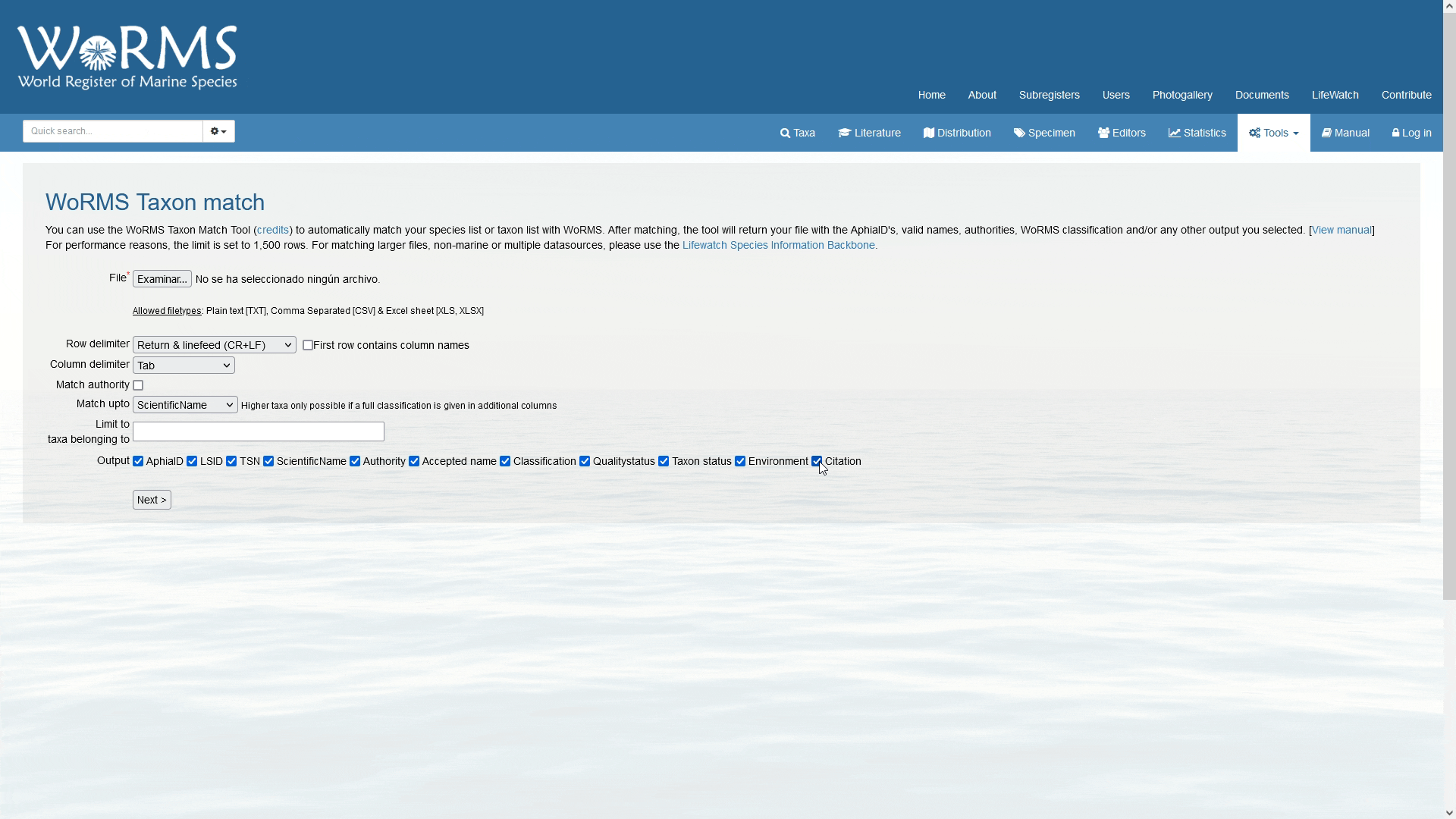
El octavo paso es escoger en una serie de botones de chequeo, cuales datos se incluirán en la salida de la consulta de coincidencia con nuestra lista taxonómica. AphiaID es el identificador único de WoRMS; LSID es el Life Science Identifier; TSN es el identificador único de ITIS; la opción de nombres válidos enumera los nombres válidos en una columna separada (esto es importante cuando su lista incluye nombres no aceptados o variaciones ortográficas). También puede solicitar recibir las banderas completas de clasificación, autoridad y estado de calidad. El formato de salida está en .txt, .xls o .SGML.
Finalmente se debe dar click en el botón  para iniciar el proceso de coincidencia.
para iniciar el proceso de coincidencia.
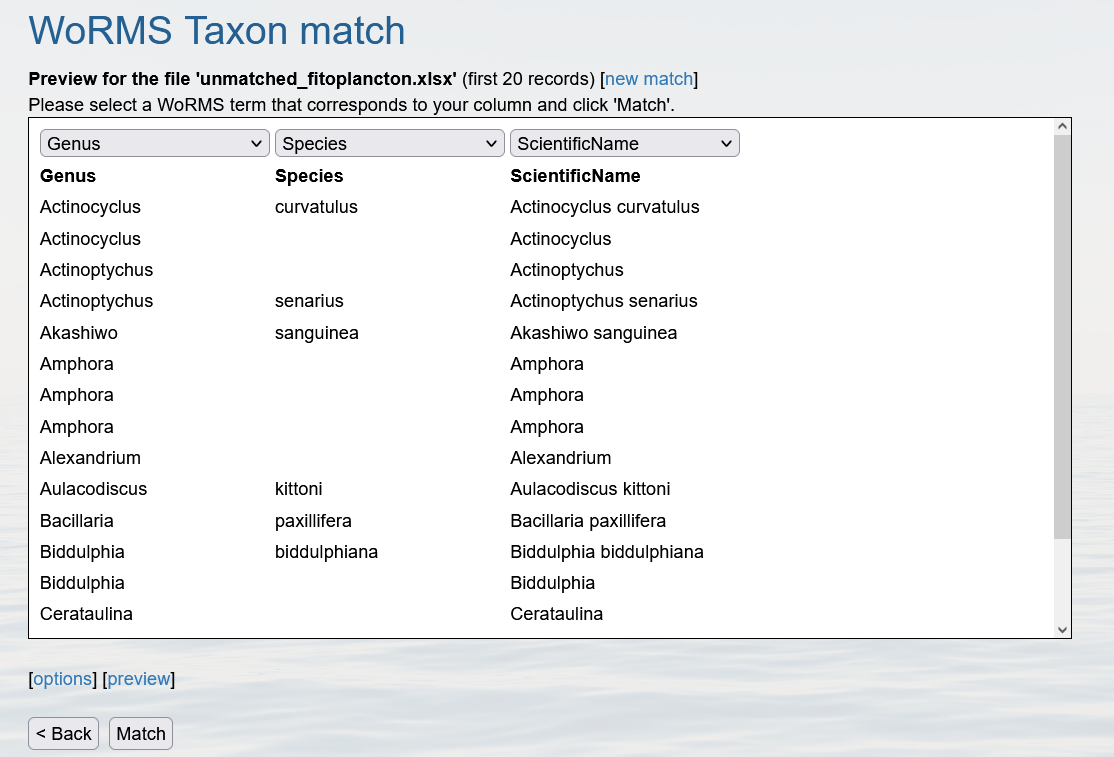
Vista preliminar de la consulta.
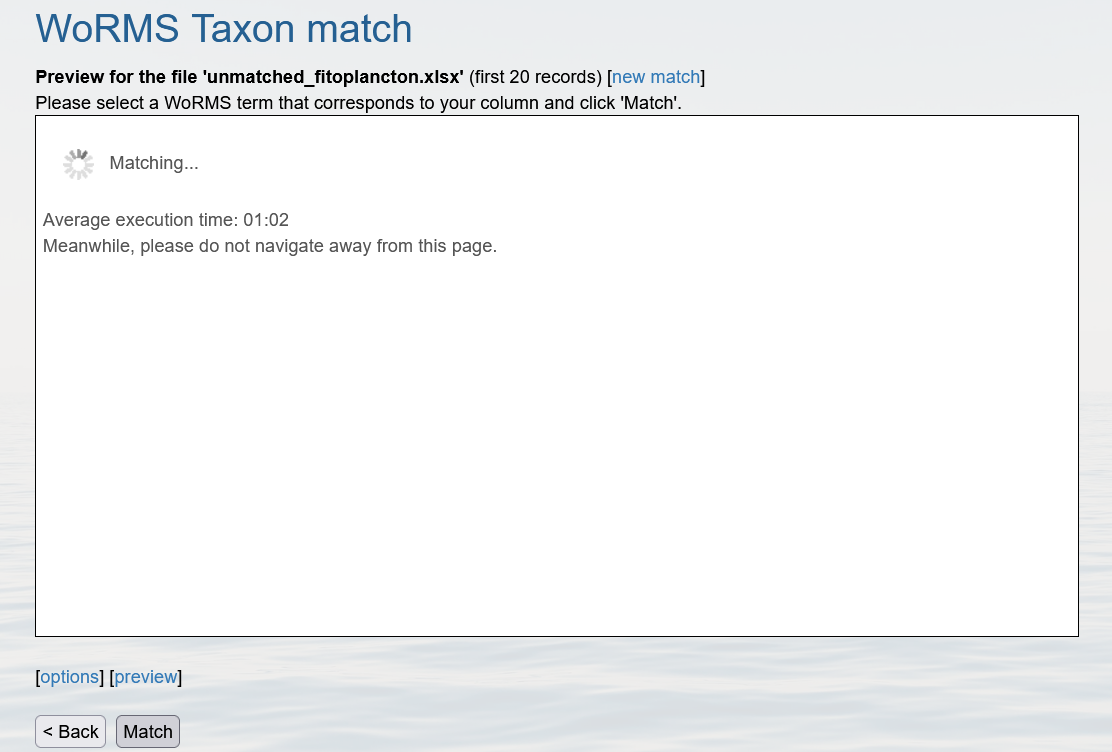
La segunda etapa de la consulta es verificar las coincidencias por niveles en la vista preliminar.
Si no existe ninguna ambigüedad se puede continuar con el proceso de coincidencia con el botón  .
.
Ejecución de proceso de coincidencia taxonómica.
En esta etapa se ejecuta el algoritmo para coincidir los datos de entrada con la base de datos taxonómica del WoRMS.
Desambiguación del resultado del proceso.
En esta etapa, la herramienta nos da la posibilidad de desambiguar cada una de las entradas de nuestro listado, ambigüedades que se pueden haber obtenido por homonimias, las cuales se tiene la posibilidad de resolver manualmente.
Existen varios tipos de coincidencia:
- exact todos los caracteres coinciden exactamente.
- subgenero_exact una coincidencia exacta, pero incluyendo el subgénero.
- phonetic suena similar a, a pesar de pequeñas diferencias en la ortografía (algoritmo soundex).
- near_1 coincidencia perfecta, excepto por un carácter. Este es un partido bastante confiable.
- near_2 buena coincidencia, excepto por dos personajes. Esto necesita un control adicional.
- near_3 buena coincidencia, excepto por tres personajes. Esto definitivamente necesita un control adicional.
- match_quarantine coincide con un nombre que se encuentra actualmente en cuarentena. En principio, cualquier nombre que se haya utilizado en la literatura no debe ponerse en cuarentena. Así que es mejor ponerse en contacto con WoRMS DMT sobre esto.
- match_deleted esta es una coincidencia con un nombre que ha sido eliminado y no hay alternativa disponible. Póngase en contacto con WoRMS DMT cuando se encuentre con esto.
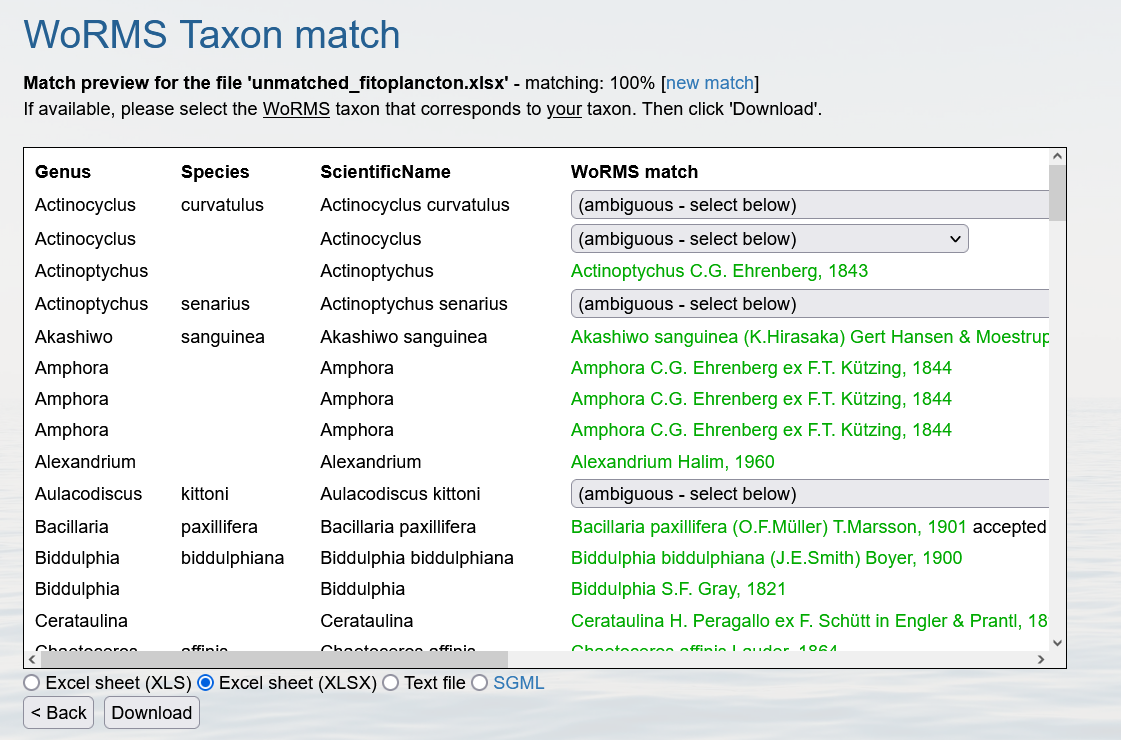
Una vez resueltas las ambigüedades, se puede descargar el archivo final en el botón  . El archivo descargado puede ser una tabla de Excel (.xls ó .xlsx), un archivo de texto (.txt) o un archivo SGML.
. El archivo descargado puede ser una tabla de Excel (.xls ó .xlsx), un archivo de texto (.txt) o un archivo SGML.
Revisión del archivo final.
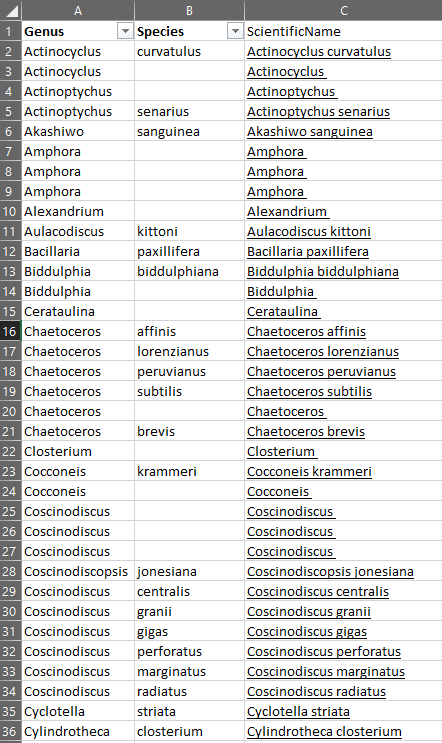
El archivo final es el resultado de las coincidencias entre el listado que se introdujo y la base de datos del WORMS. Este archivo tendrá la información taxonómica necesaria para clasificar cualquier taxón y poder alimentar con ella estándares de información de biodiversidad (e.g. DARWIN CORE).
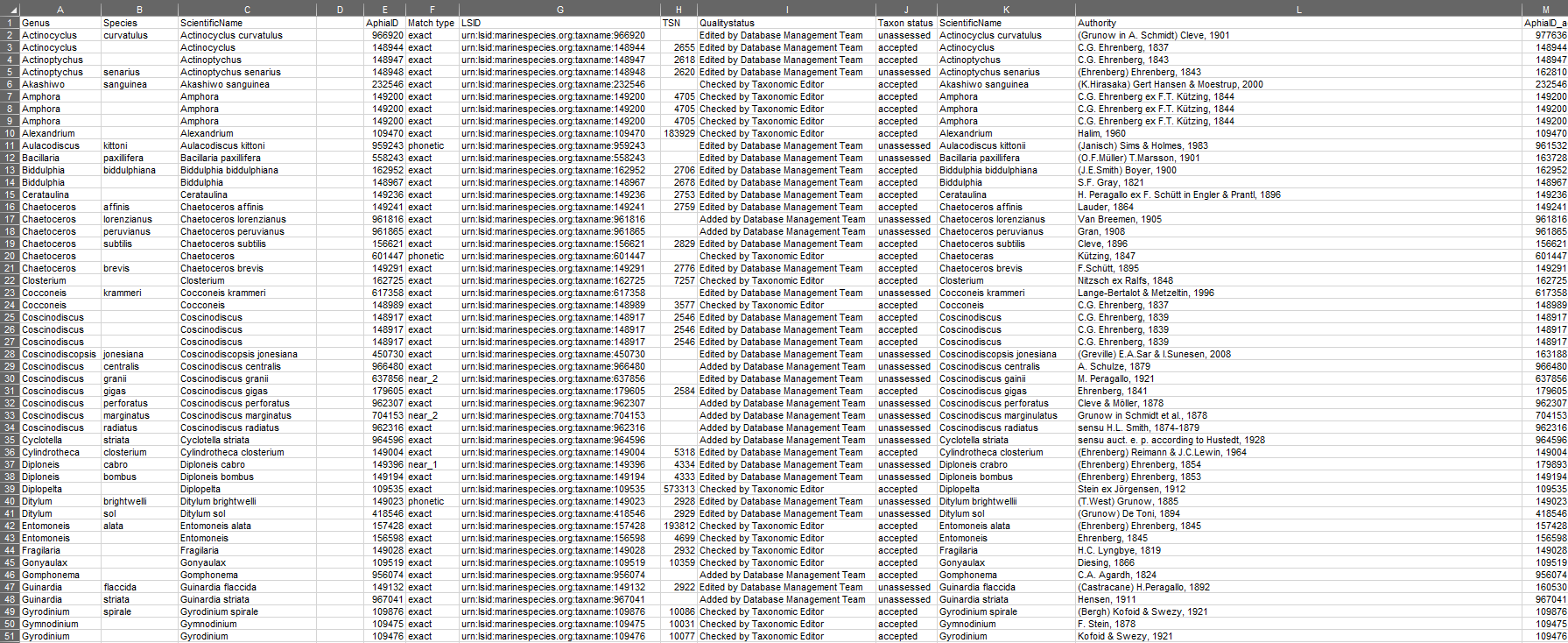
Actividad Final
Aplique el mismo procedimieto de consulta y coincidencia taxonómica con el listado taxonómico de especies de fitoplancton en formato de archívo de Excel (*.xlsx) que encontrará aquí.
El resultado obtenido debe ser similar a este: Archivo del Resultado de la consulta.